💻 Б12: Имплементација на Nussinov функција¶
Опис на барање
Да се испрограмира алгоритмот на Nussinov како функција во python која на влез ќе добива стринг од нуклеотиди, а на излезе ќе ја дава нивната секундарна структура.
Влез нека биде следната tRNA молекула:
ACCGCGGGGCGUCCGGCUCAGCUUGCCGCUGCGGAGC
GCCGCAGUAGCCGAAACUUCCGGCUCGGGCCGUGGGC
GGUCUCUAGUCGCCCCG
Алгоритам на Нусинов¶
Алгоритмот на Нусинов го решава проблемот со предвидување на секундарни РНА структури со максимизирање на базните парови.
Исто така го „решава“ проблемот со предвидување на непреодни секундарни структури. Ова се постигнува со доделување на резултат на нашата влезна структура во рамките на \(L \times L\) матрицата, \(N_{ij}\). За да го направите ова, за секој спарен комплет на нуклеотиди, ние му даваме резултат +1, а за други, 0. Потоа се обидуваме да ги зголемиме резултатите и да се повлечеме од нуклеотидите што го зголемуваат нашиот вкупен резултат. За да ги зголемиме нашите базни парови, Нусинов наведува само 4 можни правила што можеме да ги користиме при споредување на нуклеотидите.
Главните пресметки на Нусинов се рекурзивни и ги пресметуваат најдобрите секундарни структури за мали сукцесии на нуклеотиди сè додека не достигне поголеми.
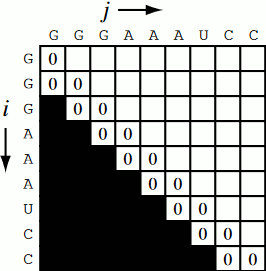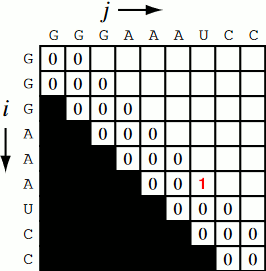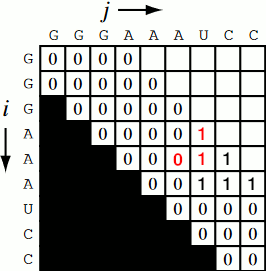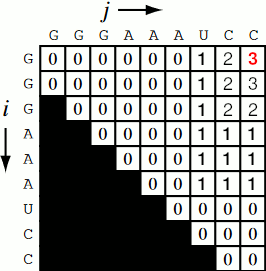
Fig. 4 Визуелн приказ за полнење на матрицата (Фаза 1)¶
Алгоритамот се состои од две фази:
Фаза на полнење на матрицата
Фаза на навраќање наназад
1. Фаза на полнење на матрицата¶
Правила:
Додадете непарирана позиција \(i\) на најдобрата подструктура за последователно ниво \(i + 1, j\)
Додадете непарирана позиција \(j\) на најдобрата подструктура за следницата \(i, j – 1\)
Додадете спарени основи \(i – j\) во најдобрата подструктура за следната \(i + j, j – 1\)
Комбинирајте две оптимални подструктури \(i, k\) и \(k + 1, j\)
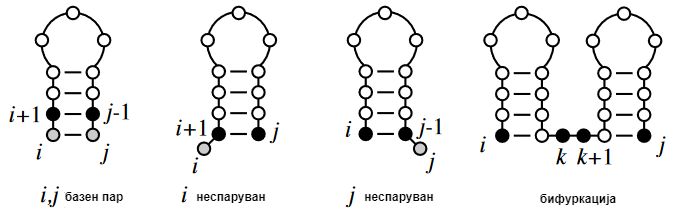
Fig. 5 Визуелен приказ на сите 4 правила¶
from Bio.Seq import Seq
import numpy as np
import pandas as pd
sample = "ACCGCGGGGCGUCCGGCUCAGCUUGCCGCUGCGGAGCGCCGCAGUAGCCGAAACUUCCGGCUCGGGCCGUGGGCGGUCUCUAGUCGCCCCG"
input_dna = Seq(sample)
output_rna = input_dna.transcribe()
print("Транскрипција")
print(f"ДНА:\n {input_dna}")
print(f"RNA:\n {output_rna}")
Транскрипција
ДНА:
ACCGCGGGGCGUCCGGCUCAGCUUGCCGCUGCGGAGCGCCGCAGUAGCCGAAACUUCCGGCUCGGGCCGUGGGCGGUCUCUAGUCGCCCCG
RNA:
ACCGCGGGGCGUCCGGCUCAGCUUGCCGCUGCGGAGCGCCGCAGUAGCCGAAACUUCCGGCUCGGGCCGUGGGCGGUCUCUAGUCGCCCCG
def fill(nm , rna):
"""
Пополнување на матрицата по Nussinov algorithm
"""
minimal_loop_length = 0
for k in range(1, len(rna)):
for i in range(len(rna) - k):
j = i + k
if j - i >= minimal_loop_length:
down = nm[i + 1][j] # 1 правило: доле
left = nm[i][j - 1] # 2 правило: лево
diag = nm[i + 1][j - 1] + couple((rna[i], rna[j])) # 3 правило: дијагонала лево долу + дали е пар или не
rc = max([nm[i][t] + nm[t + 1][j] for t in range(i, j)]) # 4 правило: комибација од две оптимални субструктури (list comprehension)
nm[i][j] = max(down, left, diag, rc) # врати го максимумот
else:
nm[i][j] = 0
return nm
def couple(pair):
"""
Врати True ако РНА нуклеотидите се парови
"""
pairs = {"A": "U", "U": "A", "G": "C", "C": "G"} # правила за комплементарност
# проверка
if pair in pairs.items():
return True
return False
def init_matrix(rna):
M = len(rna)
# иницијална матрица
nm = np.empty([M, M])
nm[:] = np.NAN
# главна дијагонала и веднаш под главна-дијагоналата
nm[range(M), range(M)] = 0
nm[range(1, len(rna)), range(len(rna) - 1)] = 0
return nm
2. Фаза за навраќање наназад¶
def traceback(nm, rna, fold, i, L):
"""
Рекурзија: Навраќање низ пополнета Nussinov матрица за да се најде оптимална РНА секундарна структура
"""
j = L
if i < j:
if nm[i][j] == nm[i + 1][j]: # 1 правило
traceback(nm, rna, fold, i + 1, j)
elif nm[i][j] == nm[i][j - 1]: # 2 правило
traceback(nm, rna, fold, i, j - 1)
elif nm[i][j] == nm[i + 1][j - 1] + couple((rna[i], rna[j])): # 3 правило
fold.append((i, j))
traceback(nm, rna, fold, i + 1, j - 1)
else:
for k in range(i + 1, j - 1):
if nm[i][j] == nm[i, k] + nm[k + 1][j]: # 4 правило
traceback(nm, rna, fold, i, k)
traceback(nm, rna, fold, k + 1, j)
break
return fold
def dot_write(rna, fold):
dot = ["." for i in range(len(rna))]
for s in fold:
dot[min(s)] = "("
dot[max(s)] = ")"
return "".join(dot)
Резултат од алгоритам¶
print(output_rna)
print(f"Должина на РНА сегмент: {len(output_rna)}")
ACCGCGGGGCGUCCGGCUCAGCUUGCCGCUGCGGAGCGCCGCAGUAGCCGAAACUUCCGGCUCGGGCCGUGGGCGGUCUCUAGUCGCCCCG
Должина на РНА сегмент: 91
rna = output_rna
nm = init_matrix(rna)
nm = fill(nm, rna)
fold = []
sec = traceback(nm, rna, fold, 0, len(rna) - 1)
res = dot_write(rna, fold)
names = [_ for _ in rna]
df = pd.DataFrame(nm, index = names, columns = names)
print(df, "\n", rna)
print(f"\nИзлез на структура во облик на точки: {res}")
A C C G C G G G G C ... A G U \
A 0.0 0.0 0.0 1.0 1.0 2.0 3.0 3.0 3.0 4.0 ... 36.0 36.0 37.0
C 0.0 0.0 0.0 1.0 1.0 2.0 3.0 3.0 3.0 4.0 ... 35.0 36.0 36.0
C NaN 0.0 0.0 1.0 1.0 2.0 2.0 2.0 2.0 3.0 ... 35.0 35.0 35.0
G NaN NaN 0.0 0.0 1.0 1.0 1.0 1.0 1.0 2.0 ... 34.0 34.0 34.0
C NaN NaN NaN 0.0 0.0 1.0 1.0 1.0 1.0 2.0 ... 34.0 34.0 34.0
.. ... ... ... ... ... ... ... ... ... ... ... ... ... ...
C NaN NaN NaN NaN NaN NaN NaN NaN NaN NaN ... NaN NaN NaN
C NaN NaN NaN NaN NaN NaN NaN NaN NaN NaN ... NaN NaN NaN
C NaN NaN NaN NaN NaN NaN NaN NaN NaN NaN ... NaN NaN NaN
C NaN NaN NaN NaN NaN NaN NaN NaN NaN NaN ... NaN NaN NaN
G NaN NaN NaN NaN NaN NaN NaN NaN NaN NaN ... NaN NaN NaN
C G C C C C G
A 37.0 38.0 38.0 39.0 40.0 41.0 41.0
C 37.0 37.0 38.0 38.0 39.0 40.0 40.0
C 36.0 36.0 37.0 38.0 38.0 39.0 39.0
G 35.0 35.0 36.0 37.0 38.0 38.0 39.0
C 35.0 35.0 36.0 37.0 37.0 38.0 38.0
.. ... ... ... ... ... ... ...
C NaN 0.0 0.0 0.0 0.0 0.0 1.0
C NaN NaN 0.0 0.0 0.0 0.0 1.0
C NaN NaN NaN 0.0 0.0 0.0 1.0
C NaN NaN NaN NaN 0.0 0.0 1.0
G NaN NaN NaN NaN NaN 0.0 0.0
[91 rows x 91 columns]
ACCGCGGGGCGUCCGGCUCAGCUUGCCGCUGCGGAGCGCCGCAGUAGCCGAAACUUCCGGCUCGGGCCGUGGGCGGUCUCUAGUCGCCCCG
Излез на структура во облик на точки: (((()))((().))(().)(()))(((((.))))(()()(((((()()((.((.)))())))())())))((((((.).(()).)))))).
3D приказ на резултатот¶
Што е FornaContainer?
FornaContainer е алатка развиена од Dash community каде се прикажува ориентиран граф кој ја репрезентира секундарната структура на нуклеински киселини (може да биде и РНА и ДНА).
Линкот до алатката е следен: https://dash-gallery.plotly.host/dash-forna-container/
Излезот од претходната ќелија го додадов како нова струтура во оваа Dash апликација и резултатот за нашето превитукување беше следно:
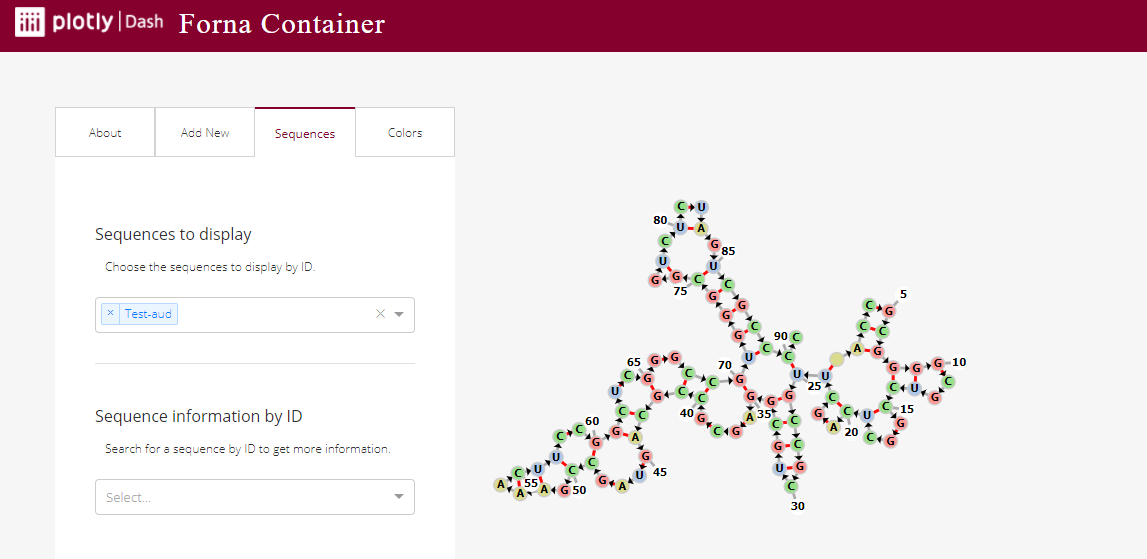
Fig. 6 Визуелен приказ на секундардната структура на нашата tRNA¶
