💻 Б13: BioPython за предвидување на секундарна структура¶
Опис на барање
Да се истражи можноста на biopython за одредување на секундарна структура опишана во поглавје 11.6.9
11.6.9 Одредување на секундарната структура¶
За оваа функционалност која библиотеката BioPython ја нуди, нужно е да се инсталира DSSP (и да се најде лиценца за истото - бесплатна за академски мејлови, https://swift.cmbi.umcn.nl/gv/dssp/). Потоа се користи DSSP класа, која мапира Residue објекти на нивната секундарна структура (и достапната секундарна површина). DSSP кодовите се прикажни во табелата 11.2. DSSP не може да се справи со повеќе модели може само еден да парсира.
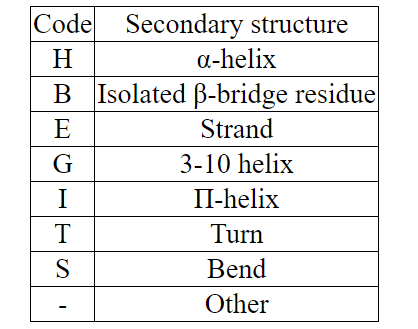
Fig. 7 DSSP кодови во Bio.PDB¶
DSSP класата исто така може да служи за пресметка на достапната површина и тоа е во поглавје 11.6.10.
Пример за приказ на PDB parser
from Bio import MissingExternalDependencyError
from Bio.PDB import PDBParser, MMCIFParser
from Bio.PDB import DSSP
# Miller (procedure similar as for the Sander values above):
p = PDBParser()
s = p.get_structure("example", "PDB/2BEG.pdb")
m = s[0]
_ = DSSP(m, "PDB/2BEG.dssp", "dssp", "Miller", "DSSP")
i = 0
with open("PDB/Miller_RASA.txt") as fh_ref:
ref_lines = fh_ref.readlines()
for chain in m:
for res in chain:
rasa_ref = float(ref_lines[i].rstrip())
rasa = float(res.xtra["EXP_DSSP_RASA"])
i += 1
import nglview as nv
from IPython.display import display
view_example = nv.show_biopython(s)
display(view_example)
